科普驿站 第三十七期
主题:核酸的剪刀——CRISPR-Cas系统
科目:生物
难度:B2
讲师:杜瑾鸿
两个含义
现在,相信你已经明白了原来CRISPR/Cas还有另外一个身份,那就是细菌的免疫系统组成成分之一。特异性感染细菌的病毒叫作噬菌体(phage),噬菌体极大地威胁了它们的生命活动。为了抵抗噬菌体,细菌进化出了多种防御措施,比如受体遮蔽、基因干扰、感染顿挫、限制修饰(R-M)系统等。以上列举的这些,是细菌的先天免疫手段,特异性低,对无论新旧的侵扰物都起反应。相比之下,CRISPR/Cas就相当于细菌的获得性免疫,特异性高,只有入侵者先前已经侵染过一次,形成记忆后(当对方再次入侵时)才能采取抵抗措施。
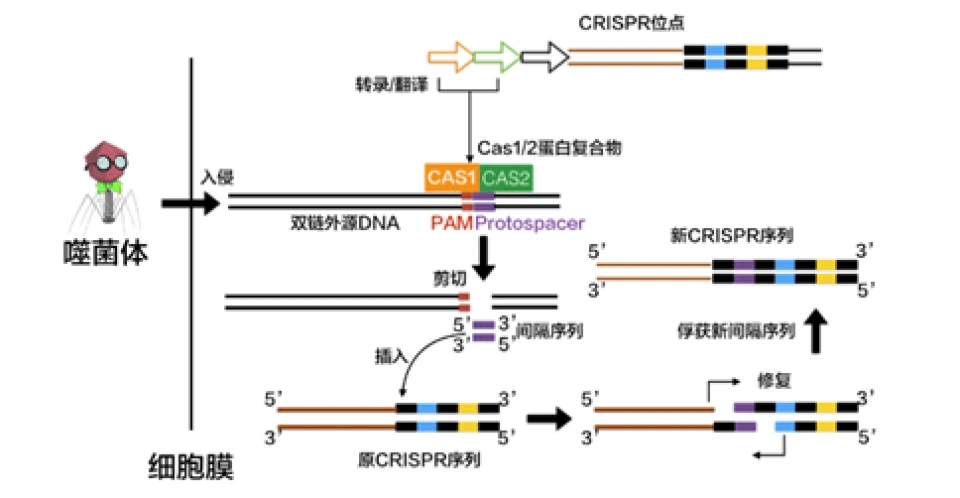
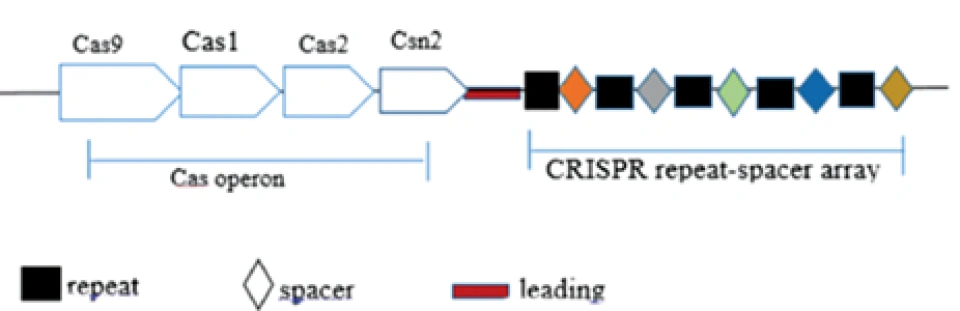
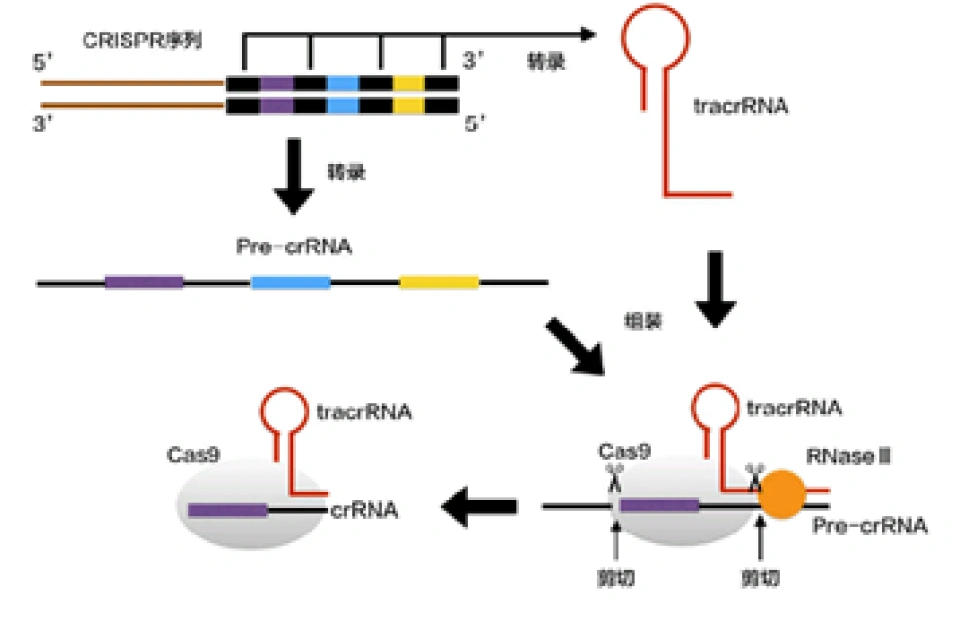
到2011年时,人们已经基本清晰地了解了CRISPR/Cas系统在DNA上的相关结构和它们的功能,即CRISPR是由一个前导序列(leader sequence,发挥类似启动子的作用,本文图中都省略),以及一个CRISPR序列(包括若干个重复序列和间隔序列)和若干个Cas基因组成的。CRISPR发挥了记忆的功能。细菌利用CRISPR/Cas来抵抗入侵者的大致机制就是:获取了入侵的噬菌体的遗传信息后,将其作为间隔序列整合进CRISPR结构,在噬菌体下次入侵时,其转录产物便能将Cas基因表达出的核酸酶定位到对应的入侵者的DNA上,核酸酶便将目标降解。
细菌具体是怎么操作这个过程的?
适应(adaptation):识别和记忆入侵者
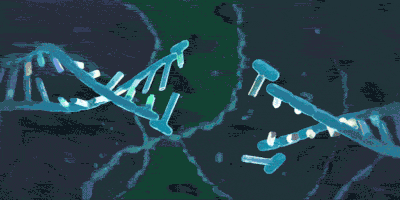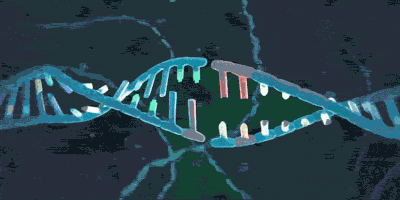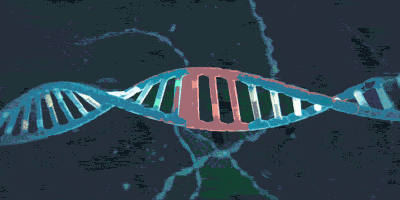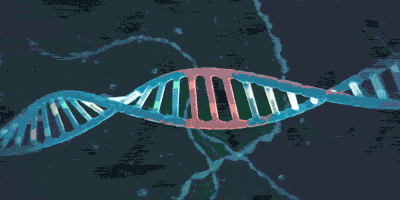
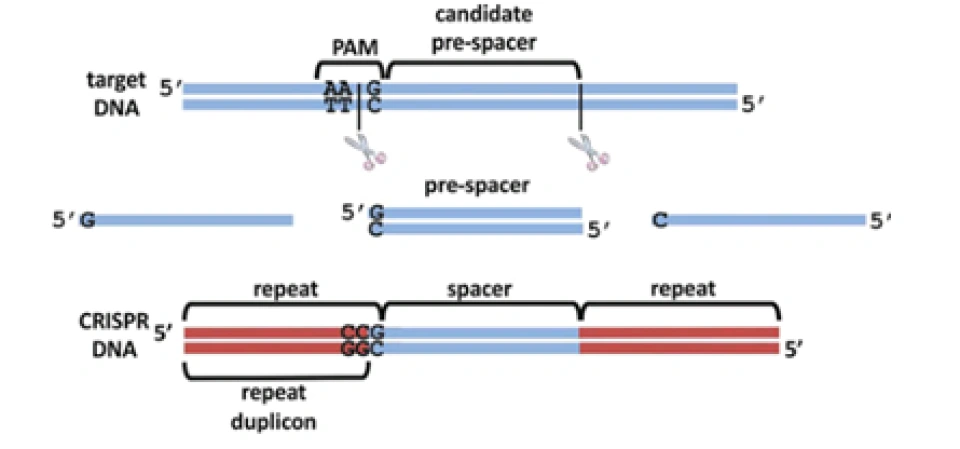
在噬菌体的DNA还是完整的的时候,未来将会被整合进CRISPR的部分称作原间隔序列(protospacer)。在紧接着原间隔序列的地方,人们发现了一个似乎被细菌当作噬菌体入侵的信号的短DNA片段,称之为“原间隔序列毗邻基序”(protospacer-adjacent motif,PAM),后者大概只有2-8个碱基。Cas在识别PAM,从而发现原间隔序列后即将其切割成小段,留下前体间隔序列(precursor spacer),并整合为CRISPR。这表明原间隔序列的选取不是随机的。每插入一个间隔序列,就要产生一个新的重复序列。通常观察到的这个操作方法是每将一段间隔序列接在原来CRISPR的末端,就复制前面一个重复序列,将新产生的重复序列与之拼接,这样一直延长;后来人们还发现另外一种插入方式,即间隔序列直接插入原来CRISPR的内部。有实验研究证实,在细菌将原间隔序列整理成间隔序列并插入CRISPR时,保留了PAM靠近原间隔序列的末端的一个脱氧核苷酸,(例如,如果在排列为-AGGGCCCTGATCGTAGCT-的序列中,AGG是PAM而GCCCTGATCGTAGCT是原间隔序列,那么被整合成间隔序列的部分就是GGCCCTGATCGTAGCT。)有人推测这跟间隔序列的精准插入的机制有关。识别与记忆的过程如图所示。
引自《【热点科普】五分钟看懂CRISPR/Cas技术》
引自Barrangou R , Oost J V D . CRISPR-Cas Systems[M]. Springer Berlin Heidelberg, 2013.p155
这一过程在几乎所有的CRISPR/Cas系统中都由Cas1和Cas2两种蛋白来完成,它们也包含在所有的CRISPR/Cas系统中。但人们对于这个过程的详细分子机制并不特别了解。而且,有的CRISPR/Cas的适应过程可能有其他蛋白参与,例如Csn2、Cas4。在II-A型CRISPR/Cas9系统中,有研究发现敲除Csn2基因导致无法形成间隔序列;有人发现Csn2蛋白形成的环状四聚体可以结合在单链的DNA末端并在其上滑动。Cas4出现在II-B和I-A/B/C/D型CRISPR/Cas系统中。人们发现在I型CRISPR/Cas系统中发现的Cas4蛋白含有一个RecB核酸外切酶结构域……这个过程的分子机制的诸细节,有待有志向的读者来探索。
表达、组装和干扰
这一过程在不同的类群的CRISPR/Cas系统中有较大差别。以CRISPR/Cas9——即II型CRISPR/Cas系统为例,其典型的DNA结构如图所示。
改编自赵宁, 李斌, 史文珍, et al. CRISPR-Cas基因编辑技术的研究进展[J]. 现代生物医学进展, 2018, 18(17):206-210.
(当噬菌体再次入侵时,)CRISPR区域转录出两种RNA分子:pre-crRNA(即crRNA的前体,就像pre-mRNA一样),这是根据CRISPR的一条链上所有的脱氧核苷酸转录而来的;tracrRNA(反式作用crRNA,trans-acting crRNA),是根据CRISPR中另一条链上的所有重复序列转录而来的,它具有发卡状(或者棒棒糖状)结构。这就意味着,tracrRNA没有形成棒棒糖结构的部分,可以与pre-crRNA对应的部分形成互补双链。由于CRISPR中掺杂的是好几种入侵者的遗传信息,而细菌只需要其中一种,所以在pre-crRNA和tracrRNA与Cas9结合后,再经过一种RNA酶——RNase III和Cas9本身的共同作用后,便可形成多种成熟的crRNA-Cas9。Cas9蛋白、crRNA与tracrRNA共同结合成为了细菌对抗入侵者的武器。
引自《【热点科普】五分钟看懂CRISPR/Cas技术》
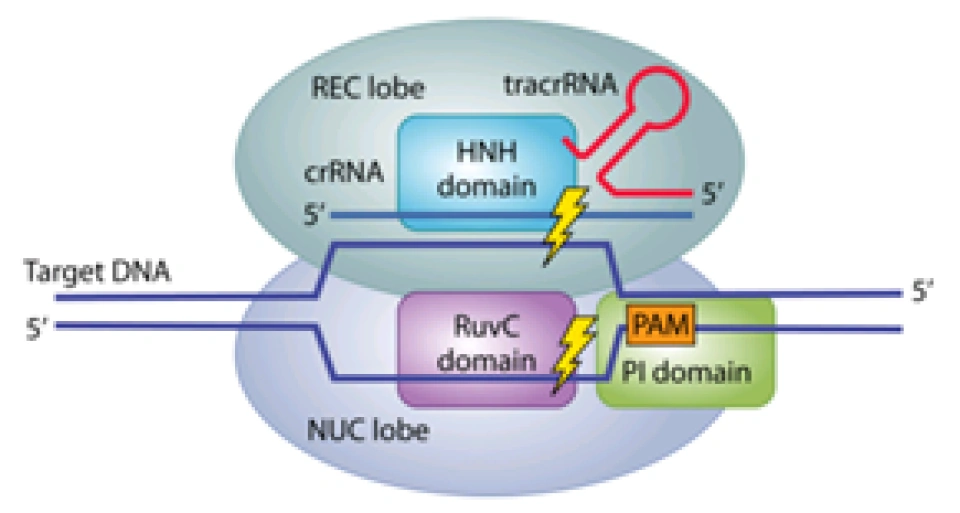
Cas9蛋白具有识别、解旋、剪切的功能,这些功能仰赖了Cas9蛋白中不同的结构域。Cas9蛋白由两个“叶”(lobe)组成,(读者可以类比肺叶、脑叶这样的称呼。此外,不妨认为是两个亚基复合组成了Cas9蛋白,这两个亚基有不同的功能。但是为什么不直接称作亚基呢?因为Cas9并不真正是由两个亚基通过简单的化学结合而构成的,而是一条肽链在经过多次折叠和加工后,形成了这样的形状,两者之间有着密切的交互,是无法分开的。)大致分别具有识别和核酸酶的功能,故分别叫作REC叶和NUC叶;有几个关键的结构域:RuvC和HNH结构域发挥核酸内切酶的功能(即“剪刀”);REC1和REC2结构域的功能是识别与结合,将Cas9蛋白锚定在目标DNA上并沿线移动;PI即PAM interacting,能识别并定位到PAM序列。酿脓链球菌(Streptococcus pyogenes)的Cas9蛋白(SpCas9)的PI结构域对应的PAM序列是NGG。
引自Ishino Y , Krupovic M , Forterre P . History of CRISPR-Cas from encounter with a mysterious repeated sequence to genome editing technology[J]. Journal of Bacteriology, 2018:JB.00580-17.
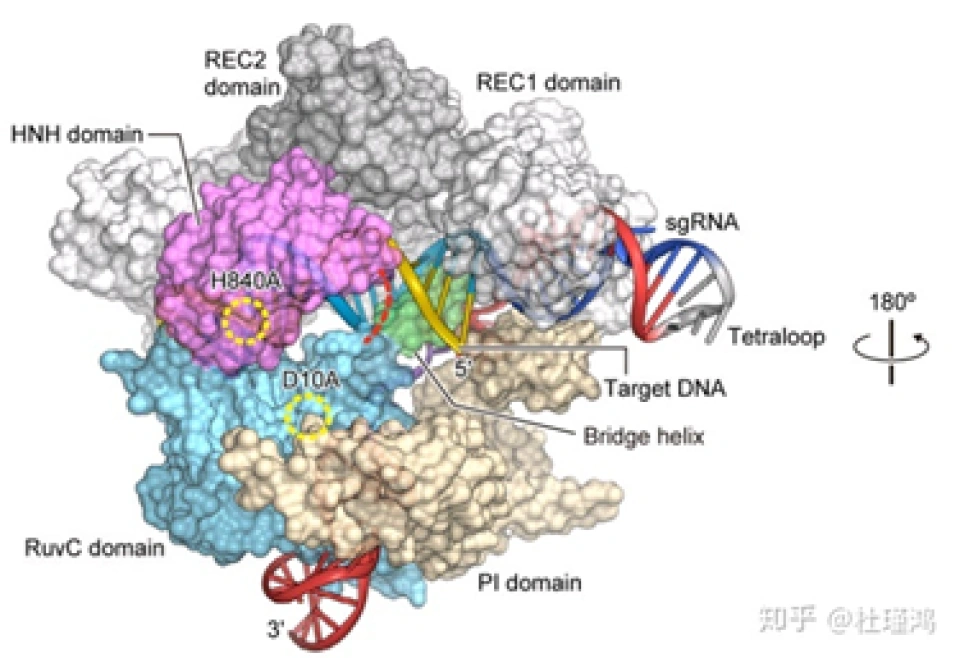
这个三聚体随后扫描外源DNA并沿着它滑动,直到crRNA中间隔序列的部分互补到原间隔序列,并且Cas9蛋白的PI结构域识别PAM——从而定位到此处,并从此处将外源DNA切开,制造出一个双链断裂(double-strand breaks,DSB),即可让外源DNA无法表达。其中,HNH结构域负责剪切的是与crRNA互补的那条链,RuvC结构域剪切的是另外一条链。如果编码Cas9蛋白的基因中,编码HNH和RuvC结构域那部分的氨基酸的碱基都发生突变,导致Cas9无法发挥切割功能而成为所谓的dead Cas9,(dCas9,dead意为“死亡的”。)这个蛋白也会锚定在目标外源DNA上,使其无法表达,最终就能达到干扰或者防御外来DNA的作用。
人们是怎么把CRISPR/Cas9改造成基因编辑技术的?
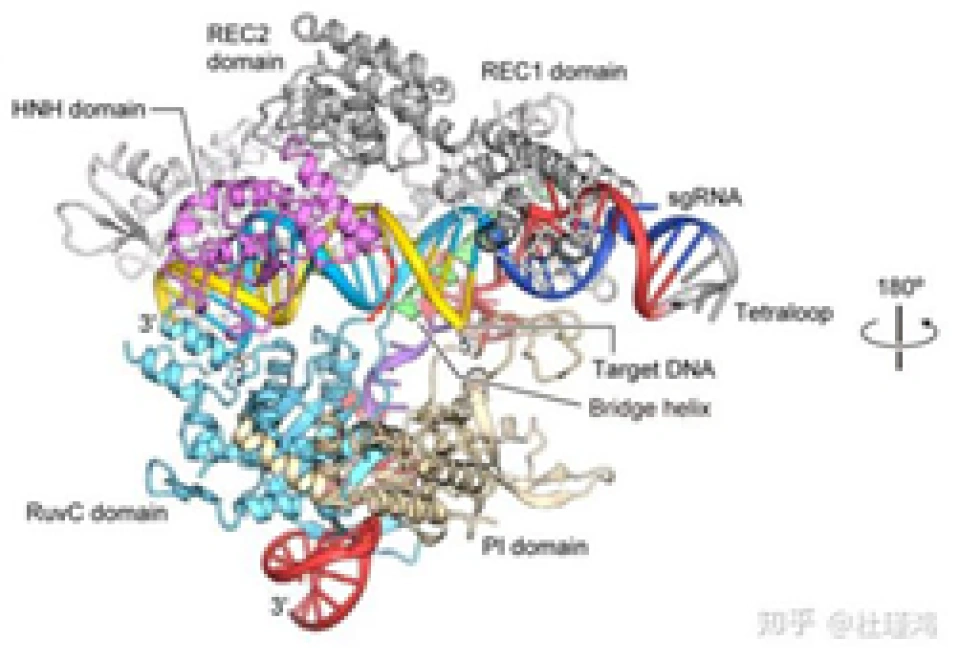
其实在介绍了细菌的CRISPR/Cas9系统运行的分子机制后,介绍CRISPR/Cas9基因编辑技术就变得简单了。为了简化操作过程,研究人员将tracrRNA-crRNA改装成了一个单股向导RNA(single guide RNA),(请读者思考“单股向导”的含义。)它与Cas9结合并靶向DNA的分子结构如图所示。sgRNA-Cas9进入细胞核并识别PAM序列和靶序列后,在其后方3个碱基的地方切断DNA,并产生平末端切口。
引自Nishimasu H , Ran F A , Hsu P D , et al. Crystal structure of Cas9 in complex with Guide RNA and target DNA[J]. Cell, 2014, 156(5):935-949,下一张图同
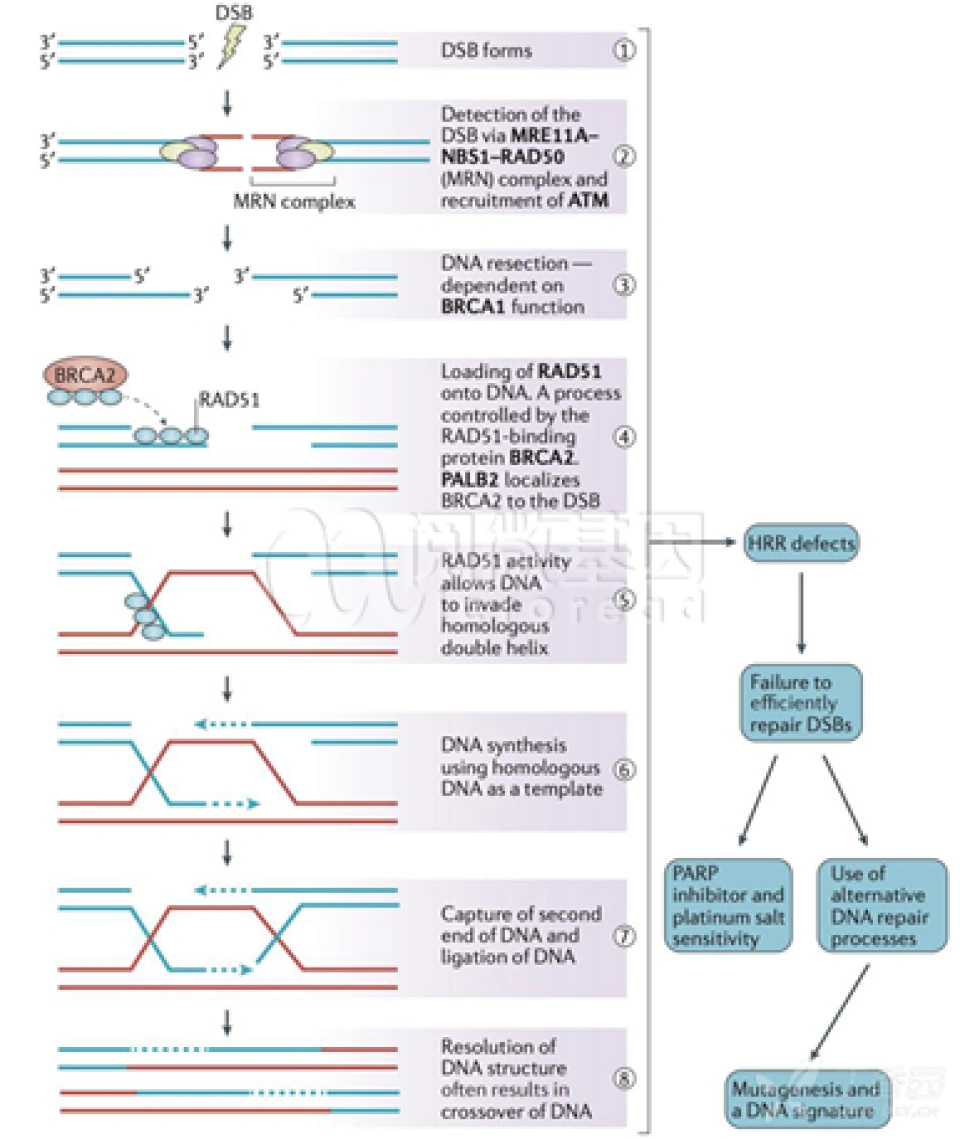
2013年,人类首次用CRISPR/Cas9在真核细胞(斑马鱼)中进行基因编辑。但我们需要考虑的是,真核细胞具有自己的方法来应对DSB(双链断裂),那就是它们的两种DNA修复机制——同源重组修复(homologous recombination,HR)和非同源末端连接(non-homologous end joining,NHEJ)。利用同源重组修复,细胞通常会以某一模板链(通常是另一条同源染色体)对缺损的另一条链进行修复(如图);而利用非同源末端连接,细胞亦会先制造黏性末端,但在之后会直接将断口接起来。这两种修复发生在细胞周期的不同时段。
来源于网络
请读者思考,如何利用它们的分子机制和发生时间的差别实现利用CRISPR/Cas9进行上面提到的几种编辑方式(删除、插入、修改等)?
当然,这两种机制都只能在分裂间期发生。分裂间期按时间顺序分为第一间隙期(G1)、合成期(S)、第二间隙期(G2)。同源重组修复不能发生在G1期。此外,要使同源重组修复特异性地发生,我们也可以利用非同源末端连接的抑制剂,比如Scr7(尽管很少有人这样做)。一方面,如果利用非同源末端连接,我们就可以删除片段或者敲低基因,如果人工构造同源重组修复的模板,我们就可以实现基因的敲入、修改、融合等。只要我们构建好了CRISPR/Cas9工具的诸零件载体,将其送入细胞核,就能按照我们的需要编辑基因了。为了将编辑工具顺利转入细胞核,科学家探索出了在载体上插入核定位信号,以及电穿孔和利用病毒整合的方法。你可以上网查询文献资料,自己了解。正是因为操作如此简单,CRISPR/Cas9便可以在几年时间里成为分子生物学技术的热门。
不过,这种机制并不是完美无缺的。
脱靶效应
sgRNA与crRNA-tracrRNA复合体一样,既存在局部双链配对的区域,也存在暴露出单链的区域(读者可以将其与tRNA比较。虽然tRNA是由单链RNA构成的,但是它的局部RNA双链配对使它形成了钥匙一样的形状)。读者可想而知,sgRNA的一部分未来通过碱基互补配对来识别要定位和切割的DNA,这一部分当然是sgRNA暴露单链的部分(之一),我们暂且称这个部分为“定位区”,称定位区与目标DNA结合的区域中,第一个碱基对与最后一个碱基对之间的部分(包含它们自己)为“结合区”,称靶DNA上的对应序列为“靶向区”(以上三个是本文临时使用的名词,不是学界公认的概念)。即我们所构造的sgRNA-Cas9复合体是通过定位区是否与目标DNA的碱基互补配对成功(以及Cas9蛋白的PI结构域是否能锚定到PAM序列)来决定是否实施切割的。
但也许与定位区能够互补配对的DNA片段在基因组里有多个,我们也许可以通过将sgRNA定位区设计得较长来解决这个问题,但太长是不行的。研究发现定位区若太长,会有一部分被剪掉,也就是等于没有用;但哪怕有局部的不配对,定位区也完全可能跟与它相似的DNA序列结合,导致我们的sgRNA-Cas9工具定位并切割错误的基因。一般来说,局部不配对有两种情况:
1.定位区与靶向区的碱基数相同,但存在碱基错配。
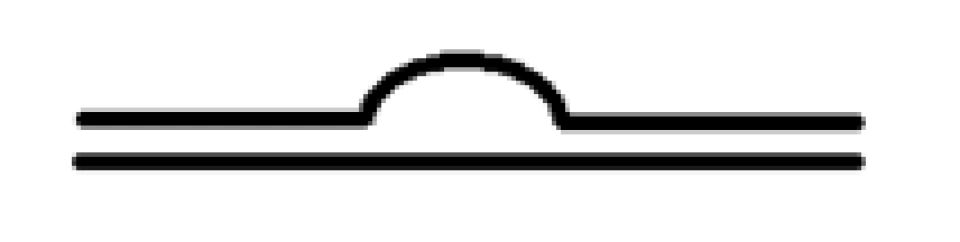
2.定位区与靶向区的碱基数不同,通过形成如图所示的凸起结构来强行配对。
你认为有哪些因素能够影响脱靶效应?
PAM序列和sgRNA的结构与长度,以及加入的sgRNA-Cas9的浓度,都可能影响脱靶效应。例如,典型的PAM序列是NGG,N表示任意碱基。试想如果能将这一碱基确定到四种碱基中的任意一种,便可大大减少sgRNA-Cas9识别的目标,故可以明显提高识别靶点的专一性。此外,如果将PAM的碱基数提高到4个、5个,也能使靶点更加精确。然而,改变PAM序列就意味着改造乃至重新设计Cas9蛋白的PI 结构域。或许这并不意味着要人工改造Cas9蛋白,也可以改变Cas9的来源,因为不同的物种的Cas9蛋白,PI结构域能识别的PAM序列不同。例如,脑膜炎奈瑟氏球菌(Neisseria meningitidis)的Cas9(NmCas9)的PAM序列有8个碱基,比酿脓链球菌Cas9(SpCas9)的NGG要严格得多。科学家还发现sgRNA(crRNA)在靠近PAM的地方存在一段种子(seed)序列,如果种子序列发生任何错配,就能导致靶点切割效率大跌甚至消失殆尽。因此合理地设计靶向种子序列也是增加特异性、降低脱靶率的方法。此外,通过改良sgRNA的结构来提高靶向效率,这也是简单易行的。现在人们已经开发出一些简单易行的sgRNA软件如http://www.genome-engineering.org/来帮助科学家设计sgRNA。
改编自尹珅, 贺桂芳, 赖方秾, et al. CRISPR/Cas9系统的脱靶效应[J]. 生物技术通报, 2016, 32(3):31-37.
错配的现象对细菌反而有利,毕竟我们无从得知原间隔序列会出现什么样的突变,即便tracrRNA-crRNA-Cas9只是强行配对识别了目标DNA,也能将其切开。但对研究者来说,出现这种情况是非常危险的,而且不仅如此,脱靶效应还可能大量存在,特别是对那些基因组十分庞大的物种——就比如拥有31.6亿个碱基的我们人类来说,对一个基因可能会存在很多个脱靶位点,让sgRNA-Cas9定位到那些脱靶位点上而不是我们想要的位置。脱靶效应导致的后果,可能是错误的表型,更麻烦的可能是假表型——删除了错误的基因,却与删除目标基因产生了相同的表型。
此外,今年6月11日,Nature Medicine上同期发表的两篇在人多能干细胞中进行相关研究发现CRISPR基因编辑造成的DNA双链断裂会激活p53,引起人多能干细胞的凋亡。p53是功能强大、十分重要的抑癌基因,CRISPR/Cas的基因编辑操作可能对被编辑的细胞起到隐形的筛选作用,而这样最后被“筛选”出来的细胞,则极有可能是存在p53功能缺陷的细胞,这就使得患者处于癌症风险之中。虽然这一分析有待直接验证,但我们仍要提高警惕。
事实上,现在已经开发出了一系列检测脱靶效应的方法,如GUIDE-seq(利用特殊的标记方法)、ChIP-seq(染色体免疫共沉淀+高通量测序)、IDLV(整合酶缺陷的慢病毒载体),还有最近新发现的DISCOVER-seq(追踪DNA修复因子MRE11)来检测CRISPR/cas9的脱靶效应或者预测可能发生脱靶效应的位点,这些方法此处不详细介绍,请读者自行了解。
脱靶效应可能对研究、实际应用,包括在医疗领域,如基因治疗等方面的应用的产生严重副作用,这正是贺建奎的实验遭到严厉谴责的另外一个原因,也是禁止以生殖为目的的对人类胚胎进行基因操作的一个出发点。
除了更加理性地设计sgRNA,科学家还有别的方法,那就是重构效应蛋白来减少或防止脱靶效应。如:
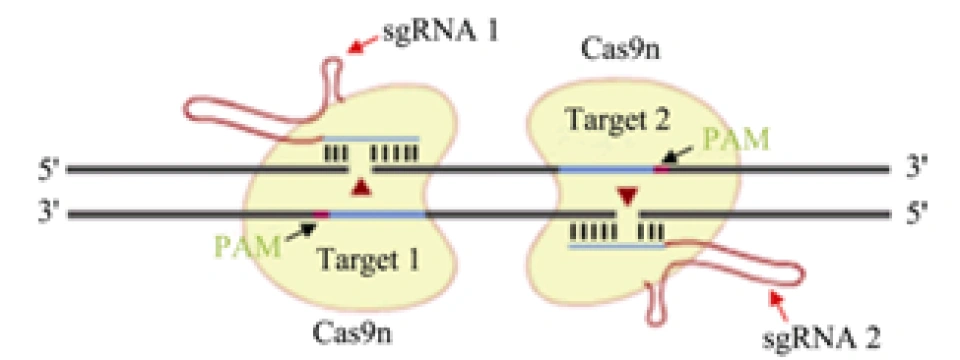
1.双切口策略(double-nicking strategy)
前面我们曾说到,Cas9蛋白具有两个核酸内切酶活性结构域。科学家构造突变型的Cas9蛋白(称为Cas9n),使它的其中一个核酸酶活性结构域被失活,故Cas9n只能切割单链。这样一来,就要设置两个朝向相反的Cas9n来产生双链断裂,并且这样断开DNA后产生的是黏性末端。此外,这样改进之后,两个sgRNA要结合不同的DNA链,PAM序列要方向相反,大大提高了特异性,因此也就提高了精确性;且产生的端口会激起细胞以非同源末端连接的方式修复DNA,若发生脱靶,两个切割位点相距很远,无法进行非同源末端连接的修复,只能利用同源模板来修复。利用这一改造过的系统,在不降低基因切割效率的前提下,将大幅降低CRISPR/Cas9的脱靶效应。
郑武, 谷峰. CRISPR/Cas9的应用及脱靶效应研究进展[J]. 遗传, 2015(10):1003-1010.,下一张图同
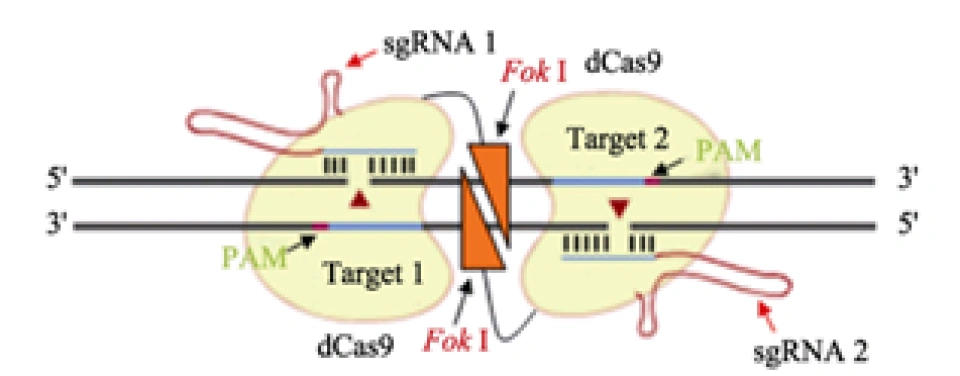
2.fCas9系统(Fok I-dCas9)
使Cas9蛋白彻底失去催化活性,使其成为dCas9,然后使其与Fok I(一种核酸酶)形成融合蛋白——Fok I-dCas9。Fok I这种核酸酶有一个特性——即需要二聚才能发挥切割功能,这使改造出的Fok I-dCas9也必须二聚才能剪切。这样一来,Cas9的实质作用就是携带sgRNA——后者正是定位到靶基因的关键。同样地,这一措施大大增加了特异性,从而大幅降低了脱靶率。
可以说,从预防脱靶效应的本质上来讲,上面两种措施是没有差别的。不过,你认为,除了增加需要配对的PAM和sgRNA碱基数,从而大幅提高特异性以外,上面两种方法还各因什么而能够降低脱靶率?或者说上面两种方法在降低脱靶率的机制上有哪些微妙的不同?
我们下期继续介绍。
参考资料:
Haft D H , Selengut J , Mongodin E F , et al. A Guild of 45 CRISPR-Associated (Cas) Protein Families and Multiple CRISPR/Cas Subtypes Exist in Prokaryotic Genomes[J]. PLoS Computational Biology, 2005, 1(6):e60.
Makarova K S , Haft D H , Barrangou R , et al. Evolution and classification of the CRISPR–Cas systems[J]. Nature Reviews Microbiology, 2011, 9(6):467-477.
在2005年的研究中,科学家首次为Cas蛋白做了分类,将45种蛋白归入鉴定出的10个亚型:核心蛋白(core proteins)、Ecoli亚型、Ypest亚型、Nmeni亚型、Dvulg亚型、Tneap亚型、Hmari亚型、Apern亚型、Mtube亚型、RAMP组件一共十个家族中。
Ecoli亚型(cse)来源于一个大肠杆菌品系:Escherichia coli K12-MG1655。
Ypest亚型(csy)来源于多个鼠疫耶尔森菌耶尔森菌(Yersinia pestis)品系。
Nmeni亚型(csn)来源于一个脑膜炎奈瑟氏球菌品系:Neisseria meningitidis Serogroup A Z2491。
Dvulg亚型(csd)来源于希尔登堡脱硫弧菌(Desulfovibrio vulgaris Hildenborough)。
Tneap亚型(cst)来源于一个新阿波罗栖热袍菌品系:Thermotoga neapolitana DSM4359。
Hmari亚型(csh)来源于一个死海盐盒菌品系:Haloarcula marismortui ATCC 43049。
Apern亚型(csa)来源于一个敏捷气热菌品系:Aeropyrum pernix K1。
Mtube亚型(csm)来源于两个结核分枝杆菌品系:Mycobacterium tuberculosis CDC1551和H37Rv。
RAMP组件(RAMP module)亚型(cmr)是一个特殊的Cas蛋白家族。
虽然在2011年进行了重新分类,但是很多蛋白根据该分类方法所赋予的名称代号却被沿用了下来,但也有很多蛋白的名字被更改。如Cas9曾被称作Csn1或Csx12。更多详细的资料可参见TIGRFAMs(



![[s-70]](https://www.bokeyuan.net/pic/image/emoji/cas/70.png)